![[*]](crossref.png) .
Dal che si deduce che per risolvere il problema del calcolo di un
.
Dal che si deduce che per risolvere il problema del calcolo di un Anche se l'algoritmo presentato in questo paragrafo non è, l'algoritmo che presentarono per la prima volta Needlman-Wunsh nel 1970 sulla rivista J. Mol. Biol., è entrato nell'uso comune riferirsi ad esso come il metodo per calcolare il costo di un allineamento globale a coppie.
Dal capitolo precedente noi abbiamo appreso che esiste una tecnica
(programmazione dinamica), che per mezzo della risoluzione dei sotto-problemi,
di cui viene mantenuta traccia, permette di risolvere il problema globale.
In particolare, si può notare che il problema dell'allineamento di due
sequenze è analogo al problema della massima sottosequenza comune,
che è stato trattato nel Capitolo ![[*]](crossref.png) .
Dal che si deduce che per risolvere il problema del calcolo di un
.
Dal che si deduce che per risolvere il problema del calcolo di un ![]() ,
non dobbiamo esplorare tutte le numerosissime soluzioni, ma ci si può affidare
alla programmazione dinamica.
,
non dobbiamo esplorare tutte le numerosissime soluzioni, ma ci si può affidare
alla programmazione dinamica.
Prima di passare alla risoluzione del nostro problema, introduciamo
la seguente notazione. Indichiamo con ![]() la sottosequenza di
la sottosequenza di
![]() che parte dalla posizione
che parte dalla posizione ![]() inclusa e termina alla posizione
inclusa e termina alla posizione ![]() inclusa.
L'analogo in Python è
inclusa.
L'analogo in Python è ![]() = s[i:j+1].
= s[i:j+1].
Definita una misura di distanza (![]() ,
, ![]() e
e ![]() ),
la soluzione del problema, si ottiene facendo le seguenti considerazioni.
Date due sequenze
),
la soluzione del problema, si ottiene facendo le seguenti considerazioni.
Date due sequenze ![]() e
e ![]() , di cui vogliamo calcolare il costo di un
allineamento ottimo, di può notare che:
se abbiamo risolto il problema di trovare gli
allineamenti ottimi tra le sotto-sequenze
, di cui vogliamo calcolare il costo di un
allineamento ottimo, di può notare che:
se abbiamo risolto il problema di trovare gli
allineamenti ottimi tra le sotto-sequenze
Per esempio se vogliamo ottenere il costo di un allineamento ottimo
tra le sequenze ![]() ="FKLA" e
="FKLA" e ![]() ="FKSTLA", il costo dell'allineamento
fino alla posizione
="FKSTLA", il costo dell'allineamento
fino alla posizione
![]() =
=![]() (FKL,FKS), sarà
(FKL,FKS), sarà
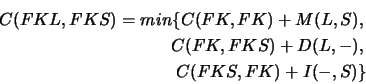
che rappresenta la scelta di minimo costo tra
Caso 1: Caso 2: Caso 3:
pos_s.....i pos_s.....i pos_s....i.
s: ...L s: ...L s: ...-
t: ...S t: ...- t: ...S
pos_t.....j pos_t....j. pos_t.....j
Siccome si vuole mantenere la simmetria tra la sequenza ![]() e quella
e quella
![]() , si pone
, si pone
![]() . Una semplice scelta è quella di
porre il costo del gap costante a crescita lineare, ossia si fissa
. Una semplice scelta è quella di
porre il costo del gap costante a crescita lineare, ossia si fissa
![]() .
In questo caso avremmo che il calcolo del costo di un allineamento diviene
.
In questo caso avremmo che il calcolo del costo di un allineamento diviene
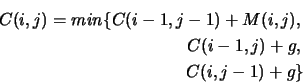
dal punto di vista grafico, significa, che dobbiamo avere una matrice
con ![]() di dimensione
di dimensione
![]() , che dobbiamo riempire
seguendo la regola
, che dobbiamo riempire
seguendo la regola
![\begin{eqnarray*}
C(i-1,j-1) &\quad &C(i-1,j) \\
\quad &_{(match)}\searrow & \downarrow^{(gap)} \\
C(i,j-1) &_{ (gap)}\rightarrow & [C(i,j)]
\end{eqnarray*}](img195.png)
Per poter procedere al computo completo abbiamo bisogno anche di quelle
che vengono chiamate condizioni al contorno.
Esse rappresentano il peso che diamo all'allineamento della sequenza ![]() contro tutti gaps
contro tutti gaps ![]() , e alla sequenza
, e alla sequenza ![]() contro tutti i gaps
contro tutti i gaps ![]() .
In pratica per l'algoritmo che stiamo considerando, vogliamo che il costo
dei singoli gaps, cresca linearmente con l'aumentare del numero. Da cui
si ha
.
In pratica per l'algoritmo che stiamo considerando, vogliamo che il costo
dei singoli gaps, cresca linearmente con l'aumentare del numero. Da cui
si ha
A questo punto siamo in grado di calcolare il costo minimo del nostro allineamento globale,
basandoci sul calcolo della distanza minima. In Figura ![[*]](crossref.png) ,
viene riportato tale algoritmo.
Come si può notare dalla Figura
,
viene riportato tale algoritmo.
Come si può notare dalla Figura ![[*]](crossref.png) ,
il costo di un allineamento ottimo è contenuto nell'ultima casella della matrice
C.
Osservando Figura
,
il costo di un allineamento ottimo è contenuto nell'ultima casella della matrice
C.
Osservando Figura ![[*]](crossref.png) e in particolare i punti #(1) e #(2),
si può verificare che la complessità computazionale
dell'algoritmo è, nello spazio occupato
e in particolare i punti #(1) e #(2),
si può verificare che la complessità computazionale
dell'algoritmo è, nello spazio occupato
Se oltre al costo, si vuole anche ottenere un allineamento, allora abbiamo
bisogno di mantenere traccia, del valore ottenuto. Questo significa che oltre
la matrice dei costi ![]() , dobbiamo utilizzare una matrice
, dobbiamo utilizzare una matrice ![]() che tiene traccia
del percorso fatto. Ossia, se la casella
che tiene traccia
del percorso fatto. Ossia, se la casella ![]() è stata aggiornata
utilizzando il match (diagonale
è stata aggiornata
utilizzando il match (diagonale ![]() ), allora dobbiamo memorizzare il percorso
nella matrice di backtrace, ponendo per esempio
), allora dobbiamo memorizzare il percorso
nella matrice di backtrace, ponendo per esempio ![]() .
Analogamente per gli altri 2 casi, che sono: colonna (
.
Analogamente per gli altri 2 casi, che sono: colonna (![]() )
) ![]() e
riga (
e
riga (![]() )
) ![]() .
L'algoritmo global_dist_al deve quindi essere modificato come riportato
in Figura
.
L'algoritmo global_dist_al deve quindi essere modificato come riportato
in Figura ![[*]](crossref.png) .
.
La procedura per poter ottenere un allineamento data la matrice di backtrace ![]() ,
la si può meglio comprendere utilizzando l'esempio riportato in Figura
,
la si può meglio comprendere utilizzando l'esempio riportato in Figura
![[*]](crossref.png) , dove si suppone di partire da una determinata
cella i,j della matrice di backtrace e di trovare nella matrice
le solite 3 possibilità.
, dove si suppone di partire da una determinata
cella i,j della matrice di backtrace e di trovare nella matrice
le solite 3 possibilità.
Un possibile algoritmo per la ricostruzione di un allineamento data
la matrice di backtrace ![]() , è descritto in Figura
, è descritto in Figura ![[*]](crossref.png) .
Esso richiede in input la matrice di backtrace B, le due sequenze seq1, seq2
e restituisce due sequenze lunghe uguali e pari alla lunghezza
dell'allineamneto specificato dalla matrice B. Ovviamente in questo
caso le due sequnze possono includere il simbolo di gap (gap_symbol='-').
.
Esso richiede in input la matrice di backtrace B, le due sequenze seq1, seq2
e restituisce due sequenze lunghe uguali e pari alla lunghezza
dell'allineamneto specificato dalla matrice B. Ovviamente in questo
caso le due sequnze possono includere il simbolo di gap (gap_symbol='-').
Come si nota facilmente dal codice (Figura ![[*]](crossref.png) ),
get_global_alignment implementa ciò che avevamo descritto
in Figura
),
get_global_alignment implementa ciò che avevamo descritto
in Figura ![[*]](crossref.png) . Ossia quando ci si muove lungo la diagonale,
si ha un 'match', ed usiamo sia un simbolo della sequenza
. Ossia quando ci si muove lungo la diagonale,
si ha un 'match', ed usiamo sia un simbolo della sequenza ![]() , che uno della sequenza
, che uno della sequenza ![]() .
Al contrario, quando ci si
muove lungo la colonna, consumiamo solo un simbolo della sequenza
.
Al contrario, quando ci si
muove lungo la colonna, consumiamo solo un simbolo della sequenza
![]() , che appaiamo con un gap. Analogamente e in modo opposto nel caso
in cui ci si muove lungo la riga (si consumano simboli in
, che appaiamo con un gap. Analogamente e in modo opposto nel caso
in cui ci si muove lungo la riga (si consumano simboli in ![]() soltanto).
soltanto).
Si può notare che in realtà l'algoritmo di Figura ![[*]](crossref.png) è più generale, e permette qualsiasi funzione di score
è più generale, e permette qualsiasi funzione di score ![]() ,
nonché, il fatto di poter calcolare sia la minima distanza (minimo tra 3 valori),
sia la massima similarità (massimo tra 3 valori).
Per poter eseguire tale algorimo abbiamo bisogno di definire queste funzioni,
ed un esempio semplice è dato in Figura
,
nonché, il fatto di poter calcolare sia la minima distanza (minimo tra 3 valori),
sia la massima similarità (massimo tra 3 valori).
Per poter eseguire tale algorimo abbiamo bisogno di definire queste funzioni,
ed un esempio semplice è dato in Figura ![[*]](crossref.png) .
Se li utilizziamo per calcolare la minima distanza tra le sequenze
"FKLA" e "FKSTLA", possiamo vedere il risultato in Tabella
.
Se li utilizziamo per calcolare la minima distanza tra le sequenze
"FKLA" e "FKSTLA", possiamo vedere il risultato in Tabella ![[*]](crossref.png) ,
in cui sono riportati i valori delle matrici e l'allineamento
corrispondente ottenuto.
,
in cui sono riportati i valori delle matrici e l'allineamento
corrispondente ottenuto.
Fino ad ora ci siamo sempre espressi dicendo che dato il minimo costo (o
massima similarità) tra due sequenze, volevamo ricostruire un allienamento
con tale punteggio e non l'allienamento con punteggio massimo.
Ciò è dovuto al fatto che, esistono casi ambigui, in cui vi possono essere
più allineamenti con lo stesso valore di distanza (similarità). In questi casi
di solito si seglie una strategia, che può essere di scegliere colonna, poi riga, ed infine
diagonale (oppure variazioni a questo ordinamento). Come esempio di ambiguità, si può
vedere il caso di della ricerca di minima distanza tra le due sequenze
ACCA, e ACA. In Tabella ![[*]](crossref.png) , sono riportate le matrici del
punteggio e quella di backtrace. Come si può notare entrambi gli allineamenti
, sono riportate le matrici del
punteggio e quella di backtrace. Come si può notare entrambi gli allineamenti
 |
| 0 | F | K | S | T | L | A | |
| 0 | 0.0 | 1.0 | 2.0 | 3.0 | 4.0 | 5.0 | 6.0 |
| F | 1.0 | 0.0 | 1.0 | 2.0 | 3.0 | 4.0 | 5.0 |
| K | 2.0 | 1.0 | 0.0 | 1.0 | 2.0 | 3.0 | 4.0 |
| L | 3.0 | 2.0 | 1.0 | 1.0 | 2.0 | 2.0 | 3.0 |
| A | 4.0 | 3.0 | 2.0 | 2.0 | 2.0 | 3.0 | 2.0 |
| 0 | F | K | S | T | L | A | |
| 0 | 0 | R | R | R | R | R | R |
| F | C | D | R | R | R | R | R |
| K | C | C | D | R | R | R | R |
| L | C | C | C | D | D | D | R |
| A | C | C | C | D | D | D | D |
| s: FK-LA |
| t: FKSTLA |
| 0 | A | C | C | A | |
| 0 | 0.0 | 1.0 | 2.0 | 3.0 | 4.0 |
| A | 1.0 | 0.0 | 1.0 | 2.0 | 3.0 |
| C | 2.0 | 1.0 | 0.0 |
|
2.0 |
| A | 3.0 | 2.0 | 1.0 | 1.0 | 1.0 |
| 0 | A | C | C | A | |
| 0 | 0 | R | R | R | R |
| A | C | D | R | R | D |
| C | C | C | D | R/D | R |
| A | C | D | C | D | D |
|
Caso Riga |
| s: AC-A |
| t: ACCA |
|
Caso Diagonale |
| s: A-CA |
| t: ACCA |