![[*]](crossref.png) avremo il risultato
avremo il risultato
In alcuni casi si preferisce penalizzare diversamente
i gap agli estremi delle sequenze da quelli inseriti all'interno,
in quanto essi potrebbero avere un significato diverso.
Per esempio se consideriamo le sequenze 'LGGV' e 'LSTWGGVK',
utilizzando l'algoritmo di allineamento globale riportato in
Figura ![[*]](crossref.png) avremo il risultato
avremo il risultato
L---GGV- LSTWGGVKcon la prima 'L' isolata, mentre se volessimo penalizzare solo i gap interni, allora potremmo avere un risultato come
---LGGV- LSTWGGVKche forse è (in questo caso) più aderente ad una realtà biologica.
Esiste un modo semplice per poter ottenere questo risultato, ed è quello di combinare l'algoritmo per la ricerca della massima similarità locale con quello della massima similarità globale. Ricordandoci infatti che con l'algoritmo local_al, riuscivamo a non penalizzare gli inizi delle sequenze azzerando la prima riga e la prima colonna. In questo modo ovunque si inizi, il costo del gap di partenza è sempre nullo indipendendentemente dal fatto che ci si trovi ad aver consumato dei simboli di una delle due sequenze (allineati con gap iniziali). A differenza però dell'allineamento locale, noi non vogliamo solo un frammento di una sequenza contro un frammento dell'altra, per cui dovremo utilizzare il calcolo del massimo tra 3 sole possibilità come nel caso di global_al, rinunciando alla possibilità di ripartire da capo come nel caso dell'allineameto locale (si confrontino max3 e max4).
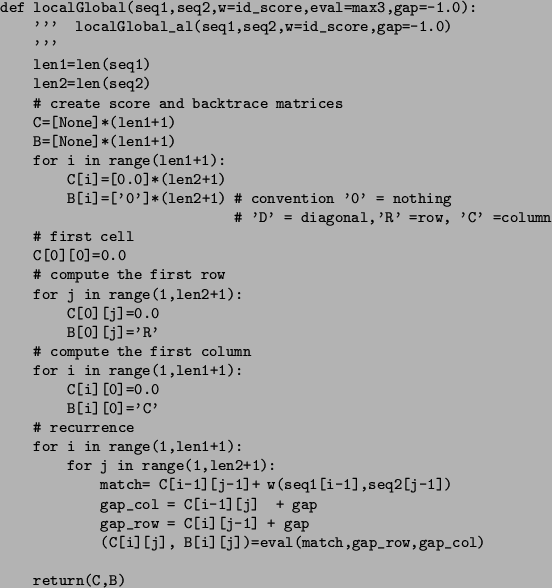
A questo punto, sappiamo che si deve utilizzare un algoritmo simile
a global_al, e con l'inizializzazione uguale a local_al,
ma ci rimane da capire cone si debbano non penalizzare i gap
al termine delle due sequenze.
Nell'allineamento globale, noi arriviamo a calcolare il punteggio massimo
contenuto nell'ultima casella della matrice. Questa posizione rappresenta
il valore massimo da noi ottenibile con le sequenze date e la nostra
misura di similarità. Ma cosa rappresentano le caselle dell'ultima
riga e dell'ultima colonna? Esse rappresentano il costo di un allineamento
globale che termina in quel determinato punto non penalizzando
i gap che sarebbero aggiunti al termine della sequenza rimanente.
Ossia nel caso fossimo nella posizione ![]() -esima dell'ultima riga (con
-esima dell'ultima riga (con
![]() dove
dove ![]() è la lunghezza della seconda sequenza), significa
che abbiamo esaurito la prima sequenza (siamo nell'ultima riga), mentre
mancano
è la lunghezza della seconda sequenza), significa
che abbiamo esaurito la prima sequenza (siamo nell'ultima riga), mentre
mancano ![]() elemetni della seconda sequenza, che quindi verrebbero
allineati con dei gap. Ovviamente il costo di questi gap non è
stato ancora calcolato, visto che non siamo all'ultima casella.
Da tutto questo possiamo dedurre che se noi partiamo dal massimo valore
che troviamo o sull'ultima riga o sull'ultima colonna,
allora abbiamo automaticamente evitato di calcolare i gap finali
delle due sequenze. Riassumendo, per non penalizziare i gap:
elemetni della seconda sequenza, che quindi verrebbero
allineati con dei gap. Ovviamente il costo di questi gap non è
stato ancora calcolato, visto che non siamo all'ultima casella.
Da tutto questo possiamo dedurre che se noi partiamo dal massimo valore
che troviamo o sull'ultima riga o sull'ultima colonna,
allora abbiamo automaticamente evitato di calcolare i gap finali
delle due sequenze. Riassumendo, per non penalizziare i gap:
![[*]](crossref.png) , dove possiamo notare che le differenze
rispetto all'algoritmo globale (Figura
, dove possiamo notare che le differenze
rispetto all'algoritmo globale (Figura ![[*]](crossref.png) ) sono solo
nell'inizializzazione della prima riga e prima colonna.
Per poter ottenere di non penalizzare gli estremi finali, dobbiamo
quindi modificare anche l'algoritmo di traceback, in modo
da partire dal massimo che ritroviamo sull'ultima riga o ultima colonna,
come riportato in Figura
) sono solo
nell'inizializzazione della prima riga e prima colonna.
Per poter ottenere di non penalizzare gli estremi finali, dobbiamo
quindi modificare anche l'algoritmo di traceback, in modo
da partire dal massimo che ritroviamo sull'ultima riga o ultima colonna,
come riportato in Figura ![[*]](crossref.png) , nella quale abbiamo fatto
uso della funzione find_maxrc che restituisce la posizione del
massimo valore dell'ultima riga o dell'ultima colonna
(Figura
, nella quale abbiamo fatto
uso della funzione find_maxrc che restituisce la posizione del
massimo valore dell'ultima riga o dell'ultima colonna
(Figura ![[*]](crossref.png) ).
).
Come esempio possiamo vedere il funzionamento dell'algoritmo descritto
controllando le matrici di score e di backtrace che vengono prodotte.
In Tabella ![[*]](crossref.png) , le sequenze 'LGGV' e 'LSTWGGVK'
vengono allineate con l'algoritmo localGlobal, e le matrici
corrispondenti sono riportate. Possiamo vedere che in questo caso
il massimo da cui partire per ricostruire l'allineamento è
nell'ultima riga e corrisponde al valore 2.0, da cui si deduce che
l'ultimo carattere della seconda sequenza sarà allineato con un gap.
La matrice di backtrace, riporta in grassetto il percorso trovato
dall'algoritmo get_localGlobal, con il corrispondente
allineamento ottenuto.
, le sequenze 'LGGV' e 'LSTWGGVK'
vengono allineate con l'algoritmo localGlobal, e le matrici
corrispondenti sono riportate. Possiamo vedere che in questo caso
il massimo da cui partire per ricostruire l'allineamento è
nell'ultima riga e corrisponde al valore 2.0, da cui si deduce che
l'ultimo carattere della seconda sequenza sarà allineato con un gap.
La matrice di backtrace, riporta in grassetto il percorso trovato
dall'algoritmo get_localGlobal, con il corrispondente
allineamento ottenuto.
 |
| 0 | L | S | T | W | G | G | V | K | |
| 0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 |
| L | 0.0 | 1.0 | 0.0 | -1.0 | -1.0 | -1.0 | -1.0 | -1.0 | -1.0 |
| G | 0.0 | 0.0 | 0.0 | -1.0 | -2.0 | 0.0 | 0.0 | -1.0 | -2.0 |
| G | 0.0 | -1.0 | -1.0 | -1.0 | -2.0 | -1.0 | 1.0 | 0.0 | -1.0 |
| V | 0.0 | -1.0 | -2.0 | -2.0 | -2.0 | -2.0 | 0.0 | 2.0 | 1.0 |
| 0 | L | S | T | W | G | G | V | K | |
| 0 | 0 | R | R | R | R | R | R | R | R |
| L | C | D | R | D | D | D | D | D | D |
| G | C | C | D | D | D | D | D | R | D |
| G | C | D | D | D | D | D | D | R | R |
| V | C | D | D | D | D | C | C | D | R |
| s: --LGGV- |
| t: LSTWGGVK |